|
(5) |
DNAの清浄さの測定(260/280ratio) |
|
|
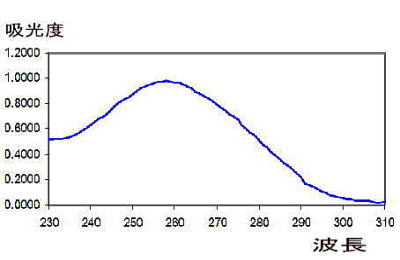
DNAやRNAは260 nmの吸光度を測定することで量(濃度)を算出することができる。260nmと280
nmの比率が純度の指標になる。260 nmの吸光度はA260またはOD260と表される。二本鎖DNAは260 nmの吸光度1が約50 μg/mlに相当し,一本鎖DNAおよRNAは260
nmの吸光度1が約33 μg/mlに相当する。この数値を基に核酸の濃度を概することができる。 |
|
|
|
|
|
※ 実際には核酸を構成している塩基(A, G, C, T)の各々の吸光度と組成が異なるめ,特に短いDNAでは組成まで考える必要がある。 |
|
|
|
|
|
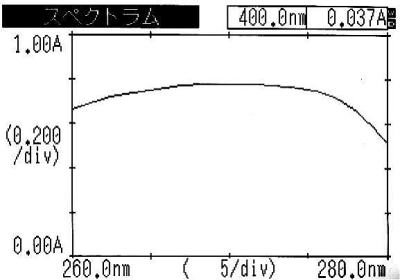
核酸の純度に関して,タンパク質の吸光を示す280 nmの吸光度との比率(A260/A280を算出することで,およその核酸の純度を知ることができる。通常,きれいにDNAがれた場合のA260/A280は1.8〜2.0を示す。これよりも低い場合はA280の値が高く,タンパク質またはフェノールの混入が考えられます。図2の場合,A260=0.66,A280=0.51より A260/A280=1.3となり,まだまだタンパク質の混入していることが考えられる。 |
|
|
|
|
|
※ 純度を上げるべきであると判断した場合,DNAの抽出(核酸の抽出)過程のフェノールによる変性作業からもう一度繰り返すことによって,徐々にタンパク質が除去されていく。 |
|
|
|
|
|
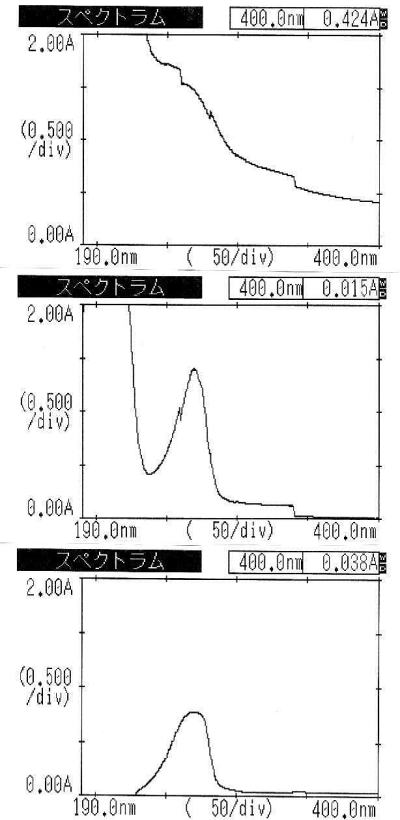
図3に -a抽出から,-bフェノール/クロロフォルム処理,-cエタノール沈殿を経るとDNAの純度が上がっていく様子を示す。 |
|
|
|
|
|
図3-a 抽出バッファーによる抽出物を遠心分離後の上清の吸収スペクトル |
|
|
図3-b 1回目の chloroformによる不純物の除去後の上清の吸収スペクトル |
|
|
図3-c 「エタノール沈殿」後の沈殿物をTE Buffer に溶解した溶液の吸収スペクトル |
|
|
 |